La ricerca del Dipartimento di Scienze e Tecnologie Alimentari dell’Università Cattolica del sacro Cuore ha utilizzato reti neurali artificiali (ANN), accoppiate all’analisi dell’impronta chimica, per il riconoscimento dell’olio prodotto a partire dalla varietà Taggiasca Ligure, raggiungendo una sensibilità prossima al 100%
La qualità dell’EVOO dipende da diversi attributi determinati geneticamente ma influenzati dalle condizioni del terreno, dalla gestione agronomica, dalla maturazione del frutto e dal tipo di produzione. In particolare, la varietà “Taggiasca Ligure” è una tipica varietà, sostanzialmente limitata alle province di Imperia e Savona, che non mostra il marchio di certificazione DOP nonostante sia riconosciuta come prodotto costoso e di alta qualità. Grazie al suo elevato valore di mercato, l’EVOO è potenzialmente esposto a frodi quali la miscelazione con altri oli o l’etichettatura fraudolenta con false dichiarazioni.
Pertanto, l’implementazione di strumenti analitici che garantiscano la tracciabilità e l’autenticità e assicurino la protezione dei consumatori e la concorrenza leale sono diventati di grande interesse sia in ambito scientifico che commerciale. A questo proposito, la metabolomica non target (impronta chimica), abbinata a statistiche multivariate e chemometria, può essere uno strumento utile per garantire la qualità e l’integrità degli alimenti.
La grande quantità di informazioni fornite dalla metabolomica richiede un adeguato trattamento dei dati, dove le reti neurali artificiali (ANN) stanno mostrando un enorme potenziale contro le frodi alimentari. Il presente lavoro combina impronta chimica e reti neurali per garantire l’autenticità dell’EVOO, utilizzando la cultivar Taggiasca come modello studio.
Luigi Lucini è laureato in Scienze e tecnologie alimentari, ha un Master in Sicurezza alimentare e il Dottorato di ricerca in Chimica, biochimica ed ecologia dei pesticidi. Professore ordinario di chimica agraria presso l’Università Cattolica del Sacro Cuore. È coordinatore della piattaforma di metabolomica e direttore del centro di ricerca interdipartimentale CRAST.
Metodologia
Campioni EVOO. Per l’analisi sono stati selezionati oli georeferenziati da diverse aree e altitudini, forniti direttamente dal consorzio dei produttori “Consorzio per la Tutela dell’Olio Extra-Vergine di Oliva DOP Riviera Ligure” (Imperia, Italia). Complessivamente, sono stati utilizzati 408 campioni, 282 dei quali erano Taggiasca originari dell’area di riferimento (province di Imperia e Savona), dalla Riviera dei Fiori ed extra-Italia. Gli altri 126 campioni erano caratterizzati principalmente dalle varietà Lavagnino, Leccino e Pignola. Da questi, 39 campioni aggiuntivi sono stati preparati in laboratorio mescolando oli Taggiasca con diverse percentuali di altre varietà (Biancolilla, Coratina, Leccino e Ogliarola – ciascuna al 10%, 50% e 80%, in volume).
Estrazione e analisi dei campioni. Il profilo dei composti fenolici e fitosterolici è stato ottenuto tramite un approccio di metabolomica basato sulla cromatografia liquida ad alta pressione (UHPLC) accoppiata a spettrometria di massa ibrida.
Statistica multivariata e reti neurali. Per quanto riguarda le reti neurali, l’architettura feed-forward multistrato è stata applicata al dataset per costruire un modello di previsione in grado di discriminare gli oli Taggiasca dagli oli non-Taggiasca basandosi sui metaboliti selezionati. Una rappresentazione semplificata dell’architettura dell’ANN in cui la rete è stata addestrata attraverso un algoritmo di retropropagazione. Infine, 32 oli Taggiasca e 30 oli non-Taggiasca indipendenti, dalla stagione di raccolta del 2022 sono stati utilizzati per convalidare il modello, e una matrice di confusione è stata creata per valutarne le prestazioni e per convalidare le differenze tramite l’analisi di transvariazione.
Risultati
È noto che diversi fattori come il clima, la varietà, la zona geografica e le pratiche agronomiche, le condizioni di estrazione e lavorazione, nonché la conservazione possono influenzare il profilo degli steroli e dei fenoli dell’olio d’oliva.
Di conseguenza, l’analisi metabolica è stata condotta per investigare in modo completo le differenze nei profili fitochimici quando: 1) si ottengono oli Taggiasca da oliveti a diverse altitudini (0-800 m s.l.m.) nella stessa area di produzione; 2) si confrontano gli oli Taggiasca con altre varietà liguri; 3) si adulterano gli oli Taggiasca mescolandoli con diverse percentuali di oli di altre varietà; 4) si confrontano gli oli d’oliva Taggiasca con altri oli d’oliva non Taggiasca.
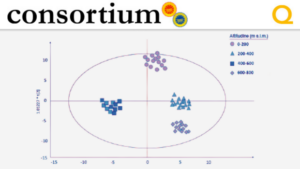
Considerando l’effetto dell’altitudine, tirosoli come l’idrossitirosolo, la demetiloleuropeina e l’aglicone dell’oleuropeina, il 3,4-DHPEA-EDA e il 3,4-DHPEA-EA erano i principali marcatori che caratterizzavano i campioni in esame, come mostrato nel grafico OPLS-DA (Fig. 1).
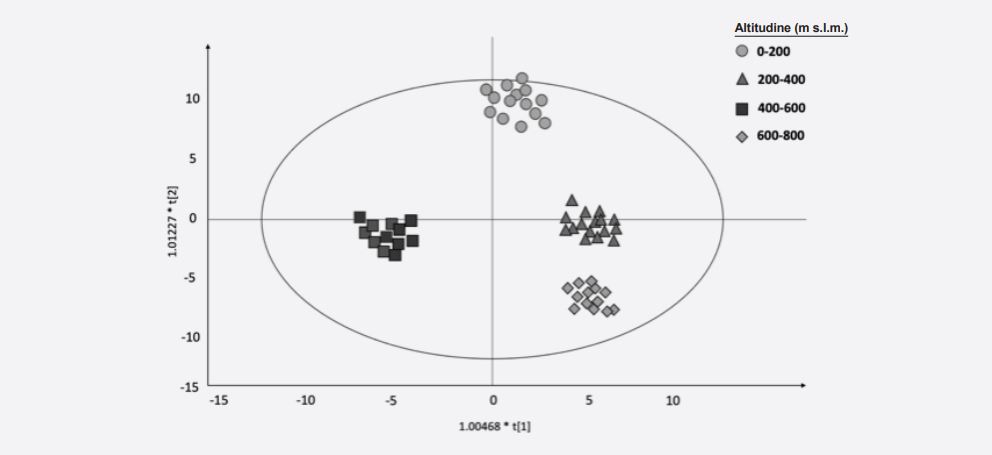
Per valutare ulteriormente l’effetto terroir, le impronte chimiche degli oli Taggiasca dell’area di coltivazione di riferimento (Imperia e Savona, definiti “autentici”) sono state confrontate con la stessa varietà proveniente da regioni diverse e con diverse varietà ma della stessa regione (Liguria). Gli oli d’oliva autentici sono stati caratterizzati da valori più elevati di lignani (pinoresinolo), mentre le altre varietà hanno registrato un maggiore contenuto di acidi fenolici, tra cui acidi benzoici, cinnamici e fenilacetici, nonché flavoni (Fig. 2).
Inoltre, oleuropeina, ligstroside e i loro agliconi, insieme a tirosolo, idrossitirosolo e loro derivati erano i composti più abbondanti negli EVOO Taggiasca Ligure di Imperia e Savona. Per quanto riguarda la miscelazione dell’EVOO Taggiasca con altri olii non-Taggiasca, i marcatori principali erano flavonoidi (16% dei marcatori), di acidi fenolici (14% dei VIP), tra cui acidi idrossibenzoici e idrossicinnamici e 32 steroli (41% dei marcatori VIP).
Il concetto di terroir è tradizionalmente utilizzato per il vino, nonostante sia chiaro che tutte le colture siano influenzate dalle condizioni pedo-climatiche. I composti fenolici e gli steroli sono metaboliti secondari con processi di biosintesi non correlati e una grande diversità chimica tra le loro sottoclassi e come altri metaboliti secondari, non sorprende che subiscano una modulazione specifica della varietà in relazione alla stagionalità, alle condizioni del suolo e del clima, e alle pratiche agronomiche. I composti fenolici sono già stati associati al concetto di “terroir” in diverse colture, compresi l’uva lo zafferano, la nocciola e il cacao sottolineando come l’impronta chimica è essenziale per catturare la diversità chimica di questi composti nei prodotti alimentari di origine vegetale.
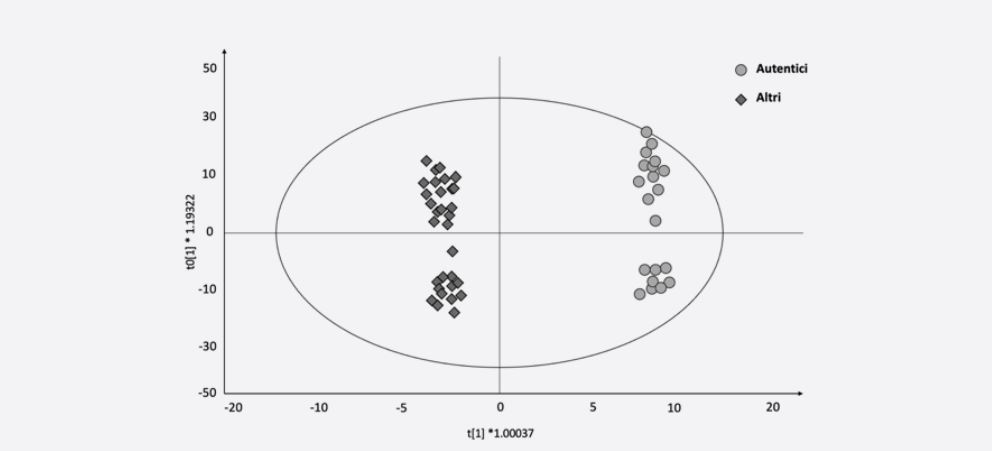
Reti neurali ANN. Il modello a reti neurali ha mostrato come, dei 62 campioni indipendenti utilizzati nella fase di validazione, 29/30 dei non autentici (Veri Negativi – TN) e 32/32 Taggiasca Ligure (varietà e territorio) siano stati classificati correttamente (Veri Positivi – TP). La sensibilità, definita come il numero di TP (32) diviso per il numero totale effettivo di campioni Taggiasca (32), è stata del 100%.
Il rapporto tra il numero di TN (29) e il numero totale effettivo di campioni non Taggiasca (30), chiamato specificità, è stato invece del 96,7%. Pertanto, ANN è stata in grado di modellare specificamente il tratto desiderato (cioè Taggiasca dalla zona di riferimento) anche in presenza di altri fattori (stagionalità, blending) in un modo che è quasi indifferente agli altri fattori confondenti.
È importante sottolineare come la modellazione delle classi come ANN si riferisca a una sola classe di interesse alla volta (nel nostro caso, Taggiasca Ligure dell’areale di riferimento). Per quanto l’impronta chimica tramite metabolomica abbia un enorme potenziale nella autenticità, indipendentemente dall’approccio chemometrico utilizzato, è essenziale un set di addestramento completo e un’appropriata convalida del modello.
Figura 1. OPLS-DA score plot di olivi a diverse altitudini. Grafico OPLS-DA basato sul profilo metabolomico degli oli ottenuti da ulivi a diverse altitudini.
Conclusioni
Gli studi sull’autenticità alimentare spesso si basano su segnali analitici che comprendono un gran numero di variabili (da centinaia a migliaia) rendendo essenziale la riduzione delle variabili e/o l’identificazione di un numero di predittori (marcatori). Il nostro lavoro si è basato sull’utilizzo della metabolomica non mirata per la cattura dell’impronta chimica (nella fattispecie, per la profilazione di fenoli e steroli) includendo un numero relativamente elevato di fattori, tutti potenzialmente influenti sul profilo dell’EVOO.
I nostri risultati hanno evidenziato che per lo sviluppo di un approccio all’integrità alimentare, un insieme così ampio di “fattori confondenti” e le dimensioni del campione, devono essere presi in considerazione nello sviluppo di un approccio di tutela dell’autenticità, affinché i risultati siano spendibili in termini realistici e non rimangano collocati all’interno della ricerca scientifica.
Da una parte, la metabolomica UHPLC/QTOF-MS abbinata al modello OPLS-DA è stata uno strumento promettente per il riconoscimento dei pattern, consentendo di verificare gli effetti della cultivar, dell’altitudine, delle condizioni pedoclimatiche e della miscelazione sull’EVOO e per la scoperta di biomarcatori (tramite l’approccio VIP). Dall’altra, l’analisi ANN ha rappresentato un modo diretto per identificare l’autentico EVOO Taggiasca, indipendentemente dal contributo specifico degli altri fattori (cultivar, altitudine, condizioni pedoclimatiche miscelazione). Pertanto, l’impronta chimica può essere utilizzata come approccio di selezione delle caratteristiche, e i marcatori “candidati” per l’autenticità essere convalidati su un ampio set di campioni.
Figura 2. Grafico OPLS-DA basato sul profilo metabolomico di oli d’oliva Taggiasca, definiti “autentici” e oli provenienti da altre cultivar liguri, definiti “altri”.
Titolo
Combining untargeted profiling of phenolics and sterols, supervised multivariate class modelling and artificial neural networks for the origin and authenticity of extra-virgin olive oil: A case study on Taggiasca LigureAutori
B. Senizza, P. Ganugi, M. Trevisan, L. LuciniFonte
Food Chemistry, Volume 404, Part A, 2023 https://doi.org/10.1016/j.foodchem.2022.134543
A cura della redazione
Fonte: Consortium 2023_04